George Whipple en 1907 (1) describió una enfermedad causada por un bacilo (actinomiceto), nunca cultivado, llamado Tropheryma Whippellii, el cual infecta al organismo por una vía desconocida. Él le llamó "lipodistrofia intestinal", pero hoy en día se conoce como enfermedad de Whipple. La enfermedad de Whipple es una entidad crónica y sistémica caracterizada por malabsorción, diarrea, fiebre, pérdida de peso, artralgias crónicas y linfadenopatía, apareciendo en ocasiones manifestaciones en el sistema nervioso central en forma de demencia, letargia y déficits motores y/o sensitivos (1-3). Manifestaciones más raras incluyen pleuritis, lesiones valvulares cardiacas, oculares u otras que pueden oscurecer la clínica y retrasar el diagnóstico antes de llegar a manifestrase por los clásicos síntomas gastrointestinales. (3,4).
Habitualmente se diagnostica por estudios histológicos, casi siempre en muestras de intestino delgado, al demostrar la existencia en la mucosa de macrófagos que contienen grandes gránulos citoplasmáticos que dan un brillante color magenta con el ácido periódico de Schiff (PAS), resistente a la diastasa (Figura 1), siendo también bacilos Gram-positivos. A menudo se aprecia una distorsión de la arquitectura de las vellosidades intestinales y dilatación de los vasos linfáticos. Estos macrófagos también pueden encontrarse en otros tejidos, tales como ganglios linfáticos, bazo, hígado o cerebro. El diagnostico, por métodos habituales, es a veces difícil y es muy difícil o imposible valorar la eficacia del tratamiento. Los estudios con microscopio electrónico han revelado la existencia de estructuras alargadas (cuerpos baciliformes) (Figura 2 y Figura 3) de 0.3 por 1.5 a 2.5 mm en el interior y cerca de los macrofagos de la lamina propia, y también en las células epiteliales y en los leucocitos polimorfonucleares. Tras el tratamiento antibiótico eficaz persisten los macrófagos en los tejidos y sólo la ME muestra la presencia de lisosomas activos con restos de bacilos sin que se encuentren bacilos viables.
Recientemente (5-7), se ha conseguido identificar al bacilo responsable de la enfermedad de Whipple mediante técnicas distintas a las tradicionales. El proceso consiste en la amplificación o el clonaje del ADN ribosómico de una población de bacterias, secuenciando y relaizando un análisis filogenético de las secuencias obtenidas (8,9). Esta aproximación ha obtenido éxito en la identificación de otras tres entidades infecciosas como son: la angiomatosis bacilar, ehrlichiosis humana, y la enfermedad de Tyzzer (9). La identificación reciente del gen 16S ribosomal, ha conducido al desarrollo de primers para la detección específica por PCR del bacilo Tropheryma Whippelli. Esta técnica puede ser ahora utilizada como estándar para el establecimiento del diagnóstico específico de la enfermedad de Whipple en pacientes en fases tempranas o de dificil diagnóstico histológico. Es más, en casos donde la histología es negativa, es posible detectar el DNA bacteriano (10).
Debido a que en el curso de la enfermedad se pueden presentar episodios de bacteriemia, el empleo de este método es útil para demostrar la presencia del Tropheryma Whippellii en células mononucleares de sangre periférica (10-12), aunque con un valor limitado debido a que no todas las muestras de enfermos, son positivas con este método (10). También es posible la detección en el caso de muestras procedentes de líquido cefalorraquídeo (13,14).
Las biopsias fueron fijadas en formol tamponado al 10% e incluidas en parafina. Seguidamente se procedió a la extracción de ADN de cortes de 4 a 6 mm de los bloques de parafina que previamente fueron desparafinados en xilol, etanol absoluto y etanol al 95%. El "pellet" una vez secado se resuspendió en tampón (50mM Tris HCl pH 8.5, 1mM EDTA y 0.5% de Tween 20) que contenía 200 mg/ml de proteinasa K, dejándose posteriormente en bano húmedo a 37 º C toda la noche. Posteriormente, se calentaron a 95 º C durante 10 minutos, almacenándose a -20 º C hasta su amplificación.
La PCR, básicamente, se llevó a cabo como sigue: los cebadores fueron W3FE (5'-GGA ATT CCA GAG ATA CGC CCC CCG CAA-3') y W4RB (5'-CGG GAT CCT GTG AGT CCC CGC CAT TAC GC-3') (Promega), midiendo los amplicones 154 bp pares de bases (bp) que corresponden a parte del gen que codifica el ARN ribosómico 16s del Tropheryma Whipellii (GenBank accesion nº M87484). Para un volumen de 50 ml de la mezcla utilizada en la reacción se emplearon, 1 ml del ADN extraido, 10mM de Tris pH 8.5, 50 mM de KCl, 0.2 mM de cada dNTP, 25 pmol de cada primer, 1,5 mM de MgCl2 y 1.2 Unidades de Taq polimerasa.
Los ciclos consistieron en una desnaturalización inicial de 5 minutos a 94 º C y 40 ciclos de amplificación de ADN. Cada ciclo consistía en 45 seg de desnaturalización a 95 º C, un "annealing" a 62 º C durante 55 seg y una extensión a 72 º C durante 55 seg, con una extensión final de 10 minutos a 72 º C.
El producto fue detectado como positivo comparando com un control positivo por electroforesis en un gel de agarosa al 2% que contenía bromuro de etidio. En cada reacción se utilizó un control positivo y uno negativo (sin anadir ADN a la reacción) (Figura 4) para excluir contaminación o resultados falsos negativos. Empleamos como controles negativos ADN de biopsias com el diagnóstico de infección por Helicobacter pylor, mycobacterium tuberculosis y mycobacterium avium.
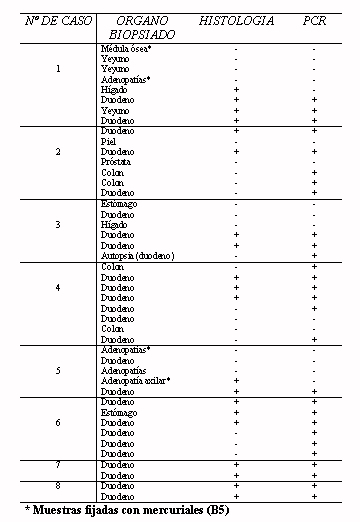
Los resultados de todas las muestras en las que se extrajo ADN están reflejados en la Tabla 1.
En 4 de las 44 muestras (9%) no se logró la extracción de ADN y en los 4 casos correspondìa a muestras fijadas con mercuriales. En 10 muestras, la histología incluyendo el estudio de microscopía electrónica, fue negativo, siendo positivo el estudio por PCR. Algunas de las muestras donde la histología era negativa y el estudio por PCR resultó positiva, corresponden a biopsias postratamiento; sin embargo la cronología exacta, así como la duración del tratamiento y la respuesta clínica y endoscópica, no están tabulados y será objeto de un estudio ulterior.
Sólo un caso donde la histología fue interpretada como positiva, la PCR fue negativa (caso 1 correspondiente a hígado).
Tabla 1. Resultados de las muestras correspondientes a pacientes con enfermedad de Whipple. Correlación con histología.

El principal objetivo de este trabajo es mostrar la posibilidad del diagnóstico de la Enfermedad de Whipple mediante la PCR. Por el momento no disponemos datos propios que correlacionen la clínica con la histología y los estudios por PCR. La mayor parte de los trabajos realizados al respecto (3,14-17), muestran una correlación buena entre la clínica y el diagnóstico por PCR.
Alguna de las biopsias en el presente trabajo que resultaron positivas por PCR y negativas por histología, son biopsias de control tras tratamiento. Esta cisrcunstancia puede deberse a dos cosas, una sería que el estudio por PCR es demasiado sensible y permite detectar el ADN bacteriano aún a pesar de una aparente curación clínica, o, puede tratarse de pacientes en los que la curación real no se ha producido y estamos ante una recaída. Por tanto, se establece la duda de aquellos pacientes que habiendo sido tratados no se negativizan van a desarrolar un curso peor, en relación a los que la PCR y la histología son negativos. A este respecto, en un trabajo (3), 7 de 12 pacientes en los que no se negativizó la PCR, presentaron recaídas y ninguno de los 5 que postratamiento eran negativos presentaron recaidas. En otro trabajo (14) siendo negativa la PCR en el intestino se observaron recaídas y manifestaciones en el sistema nervioso central en 3 pacientes de 35 estudiados, por lo que los autores proponen como método inicial de estadiaje y de monitorización testar por PCR el líquido cefalorraquídeo (LCR), a pesar de que hasta un 70% de los casos, la PCR en el LCR puede ser positiva y no presentar sintomatología neurológica (15), lo que no indica que en un futuro sea posible que se desarrolle una afectación del sistema nervioso central. En consonancia con ello se deben utilizar fármacos que atraviesen la barrerra hematoencefálica en eltratamiento de la enfermedad de Whipple dependiendo de la presencia o no de bacilos en el LCR (18).
Todos estos datos d ella literatura reflejan la falta de estudios prospectivos sobre la eficacia del test en el seguimiento de la enfermedad (10). Por otra parte es interesante el hecho de que hasta en un 35% de una muestra aleatoria de pacientes sanos se haya encontrado la presencia del bacilo, lo que sugiere que el bacilo pueda ser un comensal en la boca, lo que complica un poco más la situación, especialmente por la posibilidad de contaminación en las muestras de intestino, no así en el LCR.